抗结核化合物库
专为发现新型、高效且安全的治疗方法而设计
2 500 种化合物
结核病(TB)是全球最具威胁性的疾病之一,有可能引发下一次全球大流行。由于其传播方式和高死亡率,结核病已成为最猖獗的传染病之一。缺乏有效的治疗方法以及耐药性的迅速出现,使其成为全球公共卫生领域最紧迫的挑战之一。结核分枝杆菌(Mtb)持续发生变异,使其成为最危险且最难治疗的细菌性疾病之一。早期诊断困难以及耐药菌株的普遍存在,导致结核病极难被及时发现和治疗。因此,抗结核药物的研发至关重要,是预防未来大流行的优先事项。这需要进一步加大科学研究投入,并通过合理的药物设计来应对该疾病导致的高死亡率。
该化合物库以预装板形式提供,可快速投入使用,并为苗头化合物的后续研究提供多项支持:
- 苗头化合物及类似物补货: 可从超过440万种化合物的干粉库存中快速补充苗头化合物及其类似物,并立即进行质量控制(QC)检查。
- 药物化学(MedChem)支持: 在苗头化合物的后续验证和优化至先导化合物系列的过程中,提供专业的药物化学支持。
- 当日ADME检测: 提供当日完成的ADME(吸收、分布、代谢、排泄)测试,以帮助优先进行分子设计和合成。
典型格式
| 目录号 | 化合物数量 | 格式 | 储存形式 | 价格 |
| ATB-2500-10-Y-10 | 2 500 14块板 |
10µL@10mM DMSO溶液 |
384孔板,Greiner #781280 前2列和后2列分别留空,每板320个化合物 |
立即询价 |
| ATB-2500-50-X-10 | 2 500 53块板 |
50µL@10mM DMSO溶液 |
96孔板,Greiner #650160 第1列和第12列分别留空,每板80个化合物 |
立即询价 |
| 化合物库 & 后续跟进包: | ATB-2500-10-Y-10 化合物库,提供从干粉库存和新鲜DMSO溶液中补货的苗头化合物,并额外提供240多种来自库存的类似物,以及从REAL Space进行后续合成的类似物 | |||
* 我们很乐意以最方便您项目的格式提供我们的化合物库。请在以下我们标准的微孔板中进行选择:Greiner Bio-One 781270、784201、781280、651201 或 Echo Qualified 001-12782 (LP-0200)、001-14555 (PP-0200)、001-6969 (LP-0400)、C52621,或者您可以提供您偏好的实验器具。如需化合物混合池,我们也可以根据要求提供。
库的设计
在设计该化合物库并筛选最具前景的分子时,我们重点关注了结核分枝杆菌(Mtb)细胞壁的复杂性以及化合物穿透细胞膜所面临的困难。尽管没有采用某些通用的、固定的规则性过滤标准,但我们仍基于已发表的文献数据,以及对具有细胞活性与无细胞活性化合物的结构特征分析,尽可能剔除了所有可能在膜穿透性方面存在问题的分子。
在深入研究了结核分枝杆菌(Mtb)所特有的生物化学通路及形态学特性后,我们筛选并优先确定了一组蛋白质靶点,用于进行计算机虚拟筛选(vHTS),以挑选出具有潜在活性的化合物。总体而言,该化合物库所针对的蛋白靶点可分为两大类:与细胞壁合成相关的蛋白,以及在真核生物中不存在的、Mtb特有的必需蛋白靶点。
丙氨酸消旋酶 (Alanine racemase, Alr): 催化L-丙氨酸和D-丙氨酸之间的相互转化,D-丙氨酸是肽聚糖交联的关键组分。
dTDP-4-脱水鼠李糖 3,5-差向异构酶 (dTDP-4-dehydrorhamnose 3,5-epimerase, RmlC): 参与dTDP-L-鼠李糖的生物合成,该物质是连接阿拉伯半乳聚糖的半乳糖区域与肽聚糖分子所必需的。
烯酰基-[酰基载体蛋白]还原酶 [NADH] (Enoyl-[acyl-carrier-protein] reductase [NADH], InhA): 参与分枝菌酸的生物合成,在脂肪酸延长过程中起关键作用。
十一异戊二烯基磷酸-β-D-核糖氧化酶 (Decaprenylphosphoryl-beta-D-ribose oxidase, Dpre1): 是催化生成十一异戊二烯基磷酸阿拉伯糖(DPA)的复合物的组成部分,DPA是细胞壁阿拉伯聚糖合成的关键前体。
N-乙酰葡糖胺-1-磷酸尿苷转移酶 (N-Acetylglucosamine-1-phosphate uridyltransferase, GlmU): 催化UDP-GlcNAc生物合成的最后两步反应,对脂多糖(LPS)脂质A的合成至关重要。
莽草酸激酶 (Shikimate kinase, AroK): 利用ATP磷酸化莽草酸的3-羟基,是芳香族氨基酸和次级代谢物共同前体合成所必需的。氨基酸的生物合成是极具吸引力的药物设计方向,因其包含一系列丰富的酶家族。
β-内酰胺酶 (Beta-lactamase): 负责细菌对β-内酰胺类抗生素(如青霉素)产生耐药性。
异柠檬酸裂合酶 (Isocitrate lyase): 催化乙醛酸分流(glyoxylate shunt)的第一步反应,即从C2化合物合成C4二羧酸。
ATP酶 (ATPases, ClpC, ClpX): 作为伴侣蛋白,通过细菌蛋白酶体诱导蛋白质降解,在细菌蛋白质稳态中发挥关键作用。
Mtb调控性丝氨酸蛋白酶 (Mtb regulatory Serine Proteases, ClpP1P2): 形成细菌蛋白酶体的丝氨酸蛋白酶,负责细菌内的蛋白质稳态和降解。
肽基tRNA水解酶 (Peptidyl tRNA hydrolase, Pth): 一种Mtb特有的必需RNA水解酶,参与细菌稳态和蛋白质合成的调控。
本次虚拟高通量筛选(vHTS)流程包括:采用基于网格的方法表示结合位点,并根据文献数据(如突变实验)或配体-蛋白相互作用图谱设定约束条件,随后进行部分或完全匹配。除了依据经验性分子对接评分对化合物进行排序外,我们还对结果构象进行了人工视觉检查,以评估其结合模式的可靠性。然而,需要指出的是,针对所选靶点的参考化合物数量相对较少,且每个基于靶点的参考化合物集合内部的多样性程度较低。
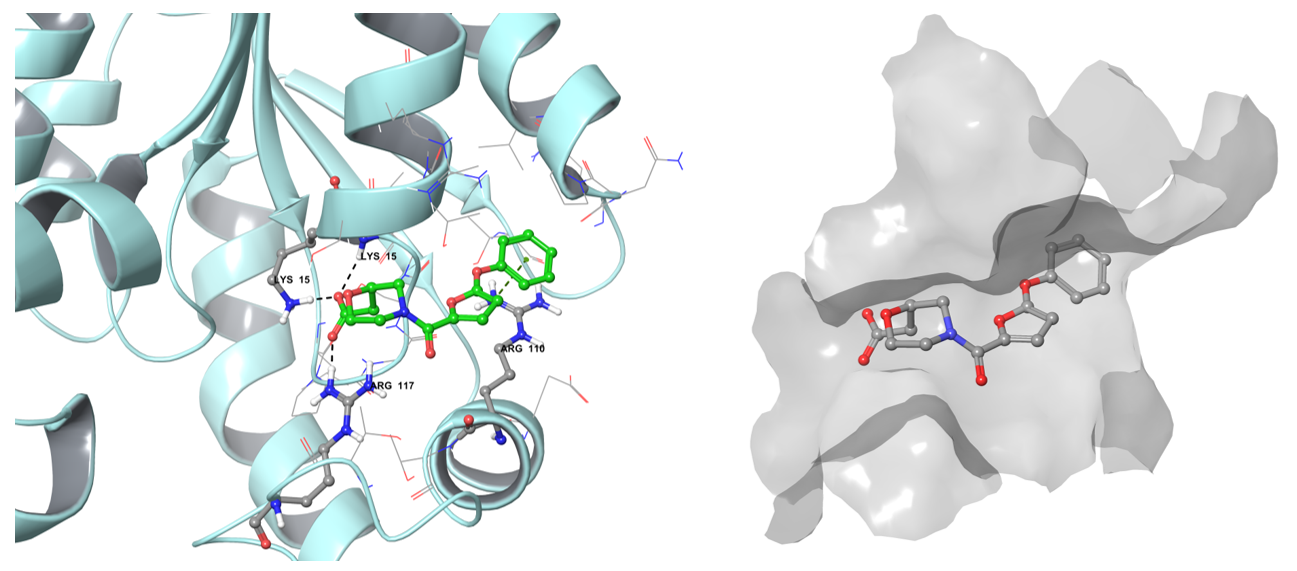
化合物 Z1603549506 在莽草酸激酶(Shikimate Kinase)结合位点中的结合模式示例。
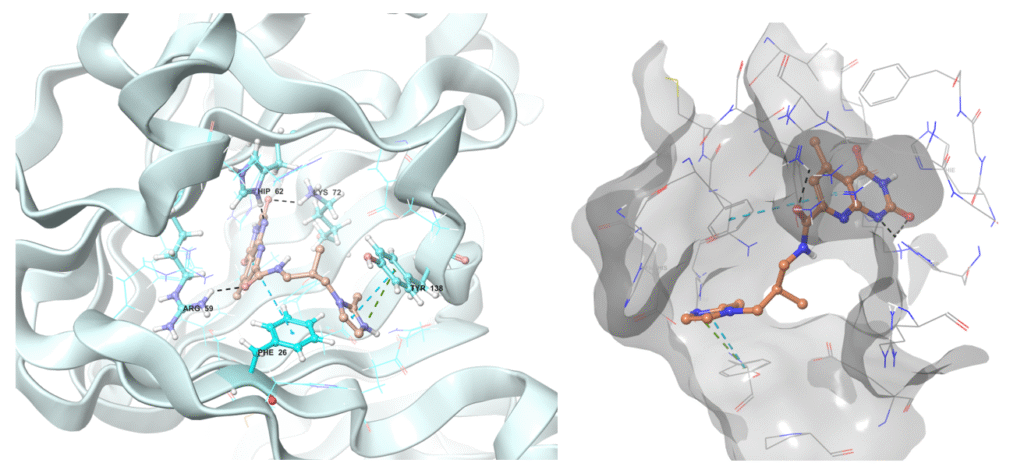
化合物 Z1313265676 与 RmlC 还原酶结合的预测构象,以蛋白质的带状图(ribbon)和形状表面(shape)表示。
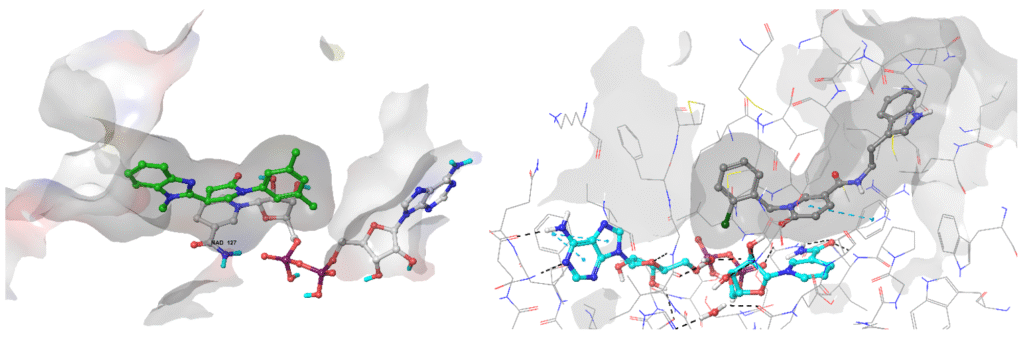
化合物 Z111780734(左)和 Z189105794 在 InhA 酶活性位点的预测结合构象示例。
参考文献
1. Consolidated Guidelines on Drug-resistant Tuberculosis Treatment
World Health Organization, 2019. Available online: https://www.who.int/tb/publications/2019/consolidated-guidelines-drug-resistant-TB-treatment/en/
2. In Silico Strategies in Tuberculosis Drug Discovery
Macalino, S. J. Y.; Billones, J. B.; Organo, V. G.; Carrillo, M. C. O. Molecules2020, 25(3), 665. doi:10.3390/molecules25030665
3. Drug Resistance Characteristics of Mycobacterium tuberculosis Isolates From Patients With Tuberculosis to 12 Antituberculous Drugs in China
Wu, X.; Yang, J.; Tan, G.; Liu, H.; Liu, Y.; Guo, Y.; Gao, R.; Wan, B.; Yu, F. Front. Cell Infect. Microbiol.2019, 9, 345. doi:10.3389/fcimb.2019.00345
4. Mycobacterium enoyl acyl carrier protein reductase (InhA): A key target for antitubercular drug discovery
Prasad, M. S.; Bhole, R. P.; Khedekar, P. B.; Chikhale, R. V. Bioorg. Chem.2021, 115, 105242. doi:10.1016/j.bioorg.2021.105242
5. Structural Basis for Inhibition of Enoyl-[Acyl Carrier Protein] Reductase (InhA) from Mycobacterium tuberculosis
de Ávila, M. B.; Bitencourt-Ferreira, G.; de Azevedo, W. F. Curr. Med. Chem.2020, 27(5), 745-759. doi:10.2174/0929867326666181203125229
6. Discovery of New and Potent InhA Inhibitors as Anti-tuberculosis Agents: Structure Based Virtual Screening Validated by Biological Assays and X-ray Crystallography
Kamsri, P.; Hanwarinroj, C.; Phusi, N.; Punkvang, A. J. Chem. Inf. Model.2019, doi:10.1021/acs.jcim.9b00918
7. New InhA Inhibitors Based on Expanded Triclosan and Di-Triclosan Analogues to Develop a New Treatment for Tuberculosis
Chetty, S.; Armstrong, T.; Sharma Kharkwal, S. Pharmaceuticals2021, 14(4), 361. doi:10.3390/ph14040361
8. Drug discovery in tuberculosis. New drug targets and antimycobacterial agents
Campaniço, A.; Moreira, R.; Lopes, F. Eur. J. Med. Chem.2018, 150, 525-545. doi:10.1016/j.ejmech.2018.03.020
9. Recent Progress in the Development of Novel Mycobacterium Cell Wall Inhibitor to Combat Drug-Resistant Tuberculosis
Belete, T. M. Microbiol. Insights2022. doi:10.1177/11786361221099878
10. Virtual screening for identifying a putative inhibitor of rmlc, a major target protein in tuberculosis disease
Sunilkumar, B.; Basheera, S. Int. J. Pharma Bio Sci.2015, 6, 616-628.
11. Spinning sugars in antigen biosynthesis: characterization of the Coxiella burnetii and Streptomyces griseus TDP-sugar epimerases
Cross, A.; Roy, S. J. Biol. Chem.2022, 298(5), 101903. doi:10.1016/j.jbc.2022.101903
12. High-resolution structures of RmlC from Streptococcus suis in complex with substrate analogs locate the active site of this class of enzyme
Dong, C.; Major, L. L.; Allen, A.; Blankenfeldt, W. Structure2003, 11(6), 715-723. doi:10.1016/s0969-2126(03)00098-4
13. Synthetic molecules as DprE1 inhibitors: A patent review
Imran, M.; A S, A.; Thabet, H. K.; Abida; Bakht, M. A. Expert Opin. Ther. Pat.2021, 31(8), 759-772. doi:10.1080/13543776.2021.1902990
14. Inhibiting Mycobacterium tuberculosis within and without
Cole, S. T. Philos. Trans. R. Soc. B2016, 371, 20150506. doi:10.1098/rstb.2015.0506
15. Advances in Key Drug Target Identification and New Drug Development for Tuberculosis
Mi, J.; Gong, W.; Wu, X. Biomed. Res. Int.2022, Article ID 5099312. doi:10.1155/2022/5099312
16. Genome-wide requirements for Mycobacterium tuberculosis adaptation and survival in macrophages
Rengarajan, J.; Bloom, B. R.; Rubin, E. J. Proc. Natl. Acad. Sci. U. S. A.2005, 102(23), 8327-8332. doi:10.1073/pnas.0503272102
17. Identification of cell wall synthesis inhibitors active against Mycobacterium tuberculosis by competitive activity-based protein profiling
Li, M.; Patel, H. V.; Cognetta, A. B. 3rd; et al. Cell Chem. Biol.2022, 29(5), 883-896.e5. doi:10.1016/j.chembiol.2021.09.002
18. Characterization of M. tuberculosis SerB2, an essential HAD-family phosphatase, reveals novel properties
Yadav, G. P.; Shree, S.; Maurya, R.; et al. PLoS One2014, 9(12), e115409. doi:10.1371/journal.pone.0115409
19. High throughput screen identifies small molecule inhibitors specific for Mycobacterium tuberculosis phosphoserine phosphatase
Arora, G.; Tiwari, P.; Mandal, R. S.; et al. J. Biol. Chem.2014, 289(36), 25149-25165. doi:10.1074/jbc.M114.597682
20. Regulatory Mechanism of Mycobacterium tuberculosis Phosphoserine Phosphatase SerB2
Grant, G. A. Biochemistry2017, 56(49), 6481-6490. doi:10.1021/acs.biochem.7b01082
21. Targeting the Serine Pathway: A Promising Approach against Tuberculosis?
Haufroid, M.; Wouters, J. Pharmaceuticals2019, 12(2), 66. doi:10.3390/ph12020066
22. Characterization of M. tuberculosis SerB2, an essential HAD-family phosphatase, reveals novel properties
Yadav, G. P.; Shree, S.; Maurya, R.; et al. PLoS One2014, 9(12), e115409. doi:10.1371/journal.pone.0115409
23. Identification of cell wall synthesis inhibitors active against Mycobacterium tuberculosis by competitive activity-based protein profiling
Li, M.; Patel, H. V.; Cognetta, A. B. 3rd; et al. Cell Chem. Biol.2022, 29(5), 883-896.e5. doi:10.1016/j.chembiol.2021.09.002
