近日,上海交通大学药学院张健老师的研究团队在论文《Fitness aligned structural modeling enables scalable virtual screening with AuroBind》,详细介绍了生成式框架 AuroBind,该框架凭借高效筛选能力,可在数千万种化合物中快速准确锁定最具新药潜力的分子。
全文篇幅较长,为方便阅读,我们已将核心内容精简提炼,呈现如下:
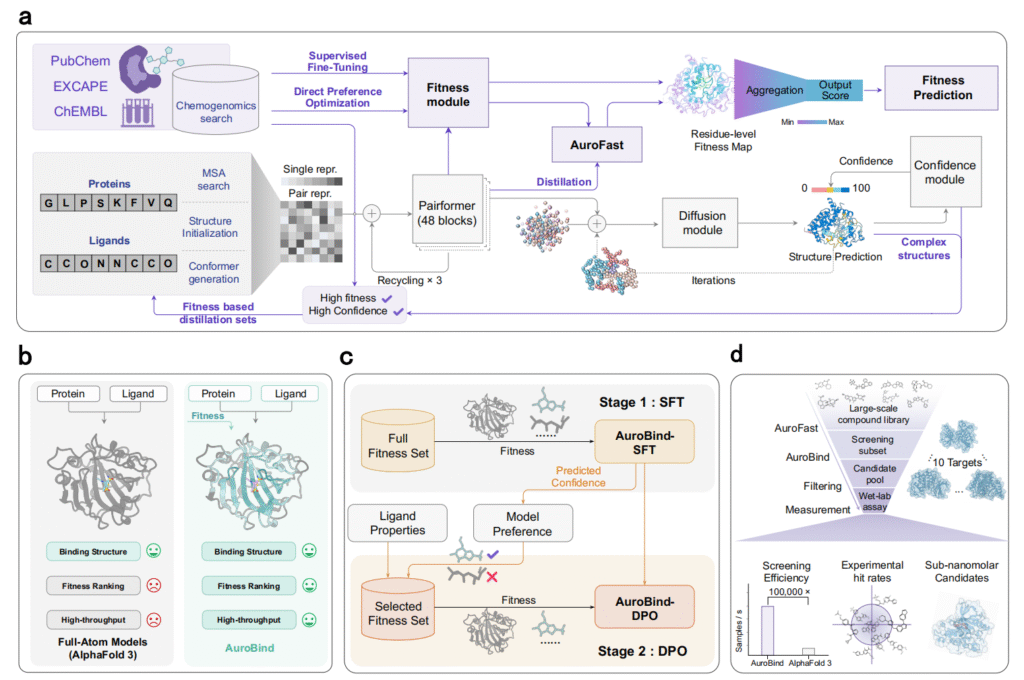
人类基因组中约 20000 个蛋白质编码基因里,仅 3%-4% 有治疗干预候选药物,药物研发面临巨大缺口。传统实验筛选耗时耗力还难精准靶向结合口袋,现有虚拟筛选方法也各有短板:基于物理的方法在特殊结合位点性能拉胯,依赖高分辨率结构;基于深度学习的方法准确性不稳定,实际效用未经验证;生成式基础模型又缺适应性感知、推理慢。
为解决这些难题,AuroBind 应运而生 —— 这是一个连接结构预测与结合适应性建模的生成式框架,堪称药物筛选的 “全能选手”。
它有三大核心优势:一是靠 33 万个蛋白质 - 配体复合物半合成数据集训练,保持 AlphaFold 3 级别的结构准确性;二是在 127 万个化学基因组测量数据(覆盖 1300 个蛋白质结构域)上微调,实现功能性适应性对齐;三是通过两阶段蒸馏得到轻量级模块 AuroFast,大幅提升筛选速度。
在性能测试中,AuroBind 表现惊艳。适应性排序上,微调后的它在 DAVIS、BindingDB 数据集的表现远超基线;筛选效率方面,AuroFast 比 AlphaFold 3 快 10 万倍以上,标准 GPU 数小时就能完成单靶点数千万化合物预筛选;结构预测准确性也超越 AlphaFold 3 和经典对接工具。
更关键的是实验验证成果。研究团队用 AuroBind 针对 10 个治疗相关靶点(涵盖受体酪氨酸激酶、表观遗传调节因子、GPCRs 等),筛选 3000 万种化合物。在双 H800 GPU 的集群上,单个靶点 24 小时内就能完成筛选,实验命中率介于 7%-69% 之间,顶级化合物效力达到亚纳摩尔至皮摩尔级别。
尤其值得一提的是,对缺乏已知配体和晶体结构的孤儿 GPCRs(如 GPR151、GPR160),AuroBind 也能精准识别有效激动剂和拮抗剂,打破了传统方法对已知信息的依赖。
AuroBind 的出现,不仅为药物研发提供了高效、精准的新工具,更有望推动更多 “不可成药” 靶点向 “可成药” 转变,为攻克疑难疾病带来新希望。

