编者按:近年来,虚拟筛选已成为加速新药研发进程的关键手段。Enamine 公司旗下的实体化合物集合 Screening Compounds 与超大虚拟化合物库 Real Space,凭借出色的可合成性及实体分子的快速交付能力,为虚拟筛选提供了强有力的支撑。本文介绍了基于 Enamine 公司 HLL-460 实体化合物库(含 46 万个化合物)开展的虚拟筛选工作 —— 在获得高阳性命中率的候选分子后,借助 Enamine 公司高效的分子快速交付服务得到18个实体化合物,经湿实验测试,成功得到了1个具有低微摩尔级活性的先导化合物。
摘要
蛋白质精氨酸甲基转移酶 5(PRMT5)可对胞质和核内蛋白质进行对称性二甲基化修饰,已被证实是重要的癌症治疗靶点。近年来,在 PRMT5 抑制剂研发方面取得了诸多进展。大多数处于临床试验阶段的 PRMT5 抑制剂主要靶向其 C 端催化结构域,而开发小分子干扰 PRMT5 与 pICLn(甲基体亚基)的蛋白质 - 蛋白质相互作用界面,对于抑制 PRMT5 同样具有重要意义。本文描述了一种基于机器学习的虚拟筛选方法,并利用这一新型流程筛选 PRMT5/pICLn 相互作用的小分子抑制剂。人工筛选出 18 种化合物进行湿实验测试,其中化合物 Z319334062 通过表面等离子体共振检测显示出对靶点的结合亲和力(解离常数 KD=21.5μM),并在患者来源的胶质母细胞瘤细胞系中剂量依赖性地抑制对称性二甲基化水平。
引言
蛋白质精氨酸甲基转移酶(PRMTs)是一类对组蛋白和非组蛋白中的精氨酸残基进行甲基化修饰的蛋白质,属于最重要的甲基化酶家族之一。这些甲基化事件调控着转录、RNA 剪接、DNA 修复、细胞周期、激素受体信号传导以及免疫反应等过程。PRMT5 作为 II 型 PRMT 酶,以 S-腺苷甲硫氨酸(SAM)为甲基供体,对蛋白质精氨酸胍基氮进行对称性二甲基化修饰,生成甲基化胍基部分和 S-腺苷高半胱氨酸,后者可被回收并重新用于甲硫氨酸的生物合成。临床和临床前研究表明,PRMT5 表达水平升高与癌症预后不良及生存率降低直接相关。因此,研发 PRMT5 抑制剂具有重要意义。
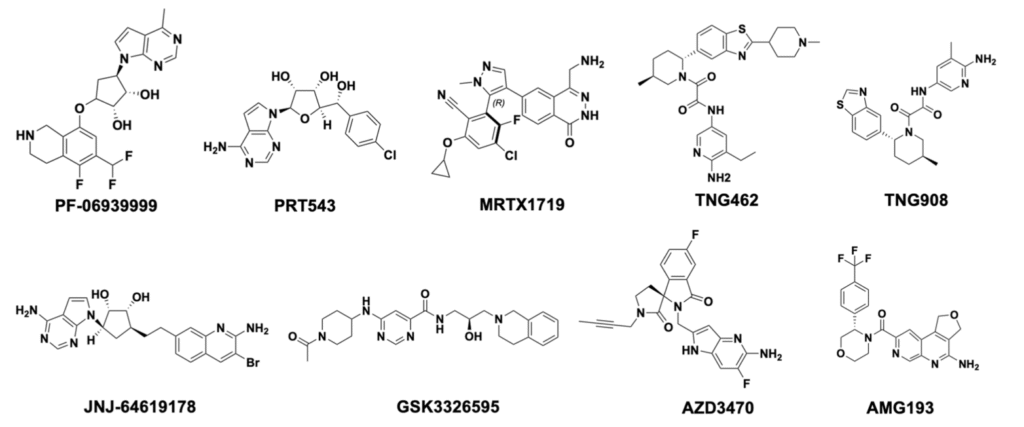
目前,大多数处于临床试验阶段的 PRMT5 抑制剂主要靶向 C 端催化结构域,可分为 SAM 竞争性抑制剂、底物竞争性抑制剂或 MTA 协同抑制剂(图 1 和图 2)。然而,McKinney等人最近发现了 PRMT5 N 端 TIM Barrel基序(PBM)与其底物衔接蛋白(pICLn、Riok1 和 COPR5)之间的新相互作用界面。这种 PBM 与底物衔接蛋白的相互作用对于招募特定底物(包括组蛋白和剪接体复合物)至 PRMT5 甲基化位点是必需的。研究人员已开发出两种先导化合物——共价抑制剂 BRD0639 和环肽(peptide 50)来干扰这种相互作用。两种分子均显示出生物化学活性和靶向细胞活性,为开发新型 PRMT5 抑制剂提供了探索方向。
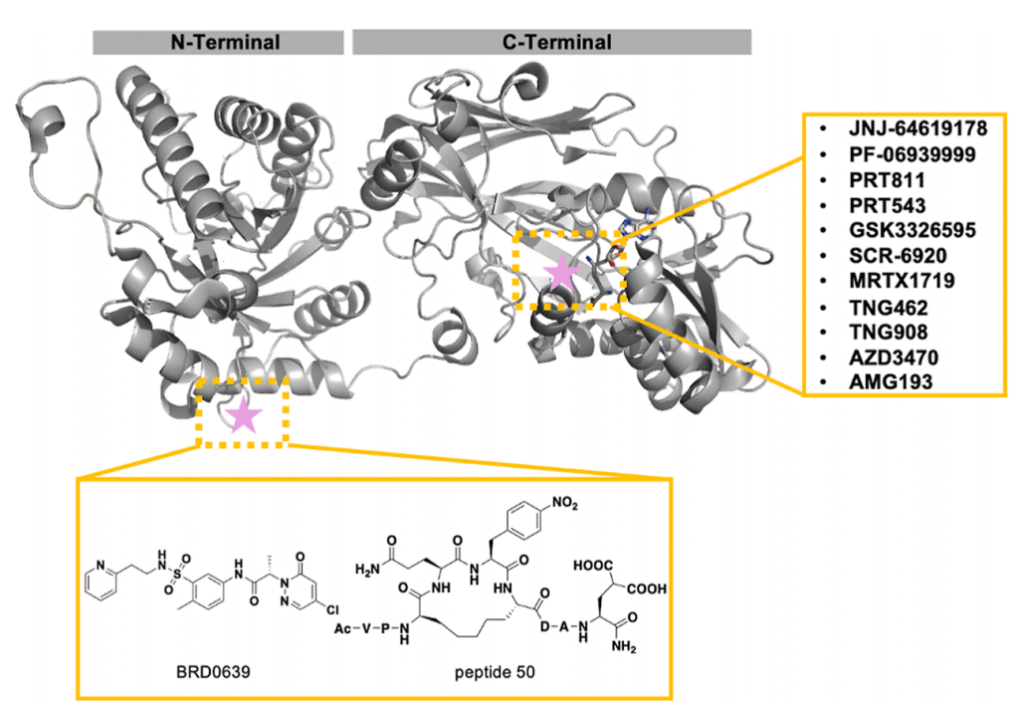
同时,Beketova等人证实,在去势抵抗性前列腺癌(CRPC)中,抑制 PRMT5/pICln 相互作用可避免雄激素受体(AR)及 AR 剪接变体的再激活,这为 CRPC 治疗提供了新视角。在先前的机制研究基础上,研究人员解析了全长 PRMT5:MEP50 与 pICLn 的冷冻电镜结构(未发表结果)。尽管由于 pICLn 的高度灵活性,大部分残基不清晰,但发现残基 205-234 与 PRMT5 N 端 TIM Barrel结构域结合明确。令人惊讶的是,还发现了一个次级结合口袋(图 3a)。基于冷冻电镜结构,pICLn205-213 的短环与 PRMT5 形成了范德华力和氢键相互作用网络。pICLn 的 Ile211 疏水侧链恰好嵌入由 PRMT5 的 Phe40、Val83、Pro120、Ala121 和 Trp152 侧链组成的疏水口袋中(图 3b)。与 PBM 位点的平坦表面和浅沟不同,这个新定义的口袋疏水性更强、更深,适合非共价小分子抑制剂结合。因此,本研究旨在利用基于机器学习的虚拟筛选流程,识别通过靶向这一新定义口袋来破坏 PRMT5/pICLn 相互作用的配体。
结果和讨论
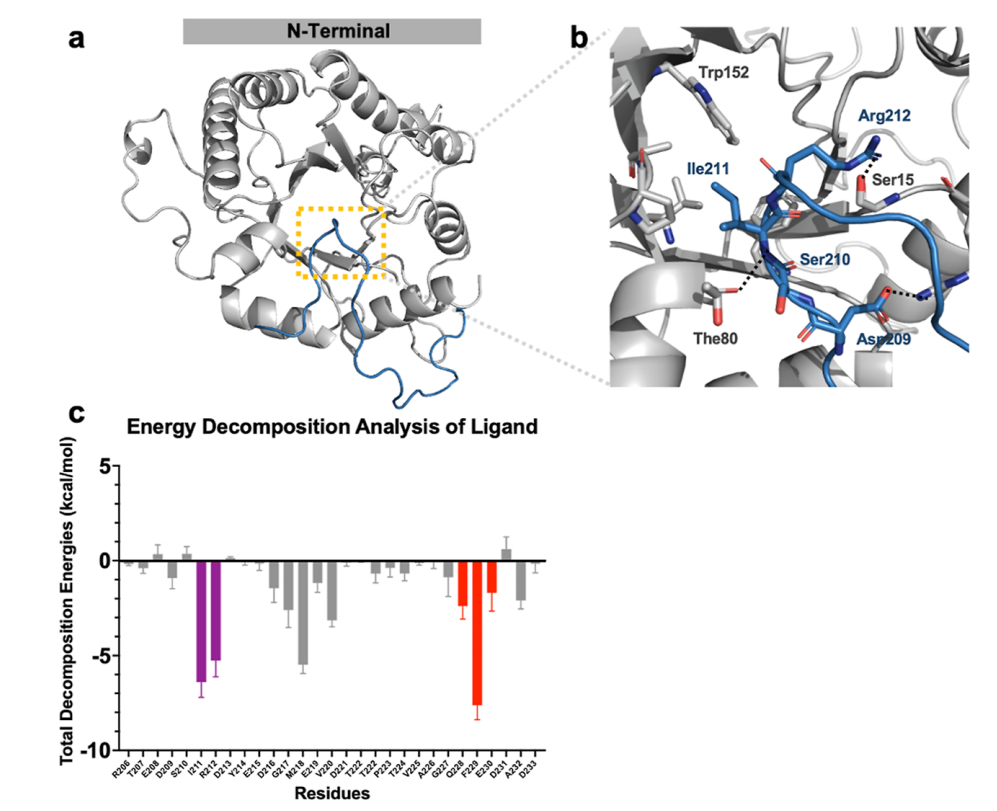
为进一步验证该疏水口袋的关键残基,本研究通过 MMPBSA 进行单残基结合能分解计算,以评估系统中单个残基的结合能贡献。首先,人工截断(PRMT5)4 (MEP50)4 (pICLn)4 冷冻电镜结构,仅保留 PRMT5 N 端 TIM Barrel(残基 1-292)和 pICLn(残基 205-234),然后对截断结构进行 200ns 的分子动力学模拟。结果显示,pICLn(残基 205-234)肽段中有两组残基对整体结合相互作用贡献最大:新定义位点的 Ile211 和 Arg212;PBM 位点的 Gln228、Phe229 和 Glu230(图3c)。因此,除 PBM 位点外,pICLn205-213 也参与了 PRMT5/pICLn 相互作用,这与Krzyzanowski等人的研究结果一致。
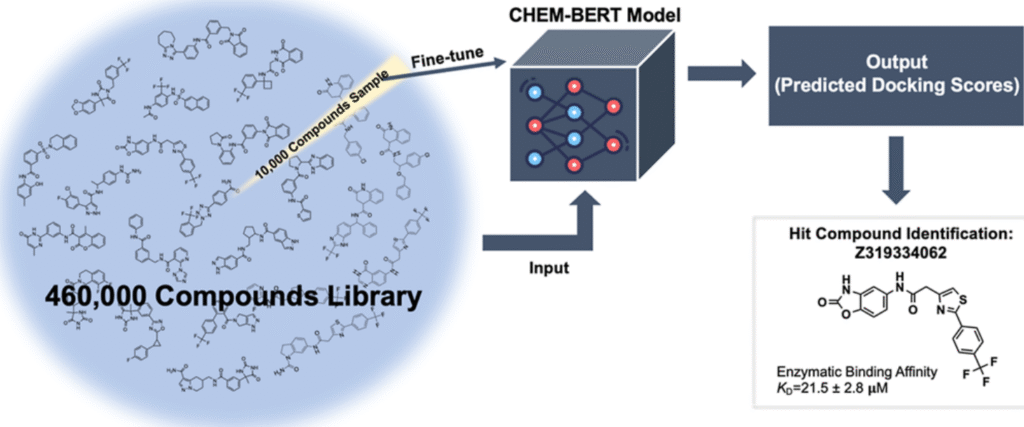
在验证配体结合位点后,本研究应用基于机器学习的虚拟筛选方法(图4)来筛选结合这一新定义口袋的小分子抑制剂。首先,从 Enamine HLL-460 化合物库(46 万个化合物)中随机选取 10,000 个多样化合物,使用 AutoDock Vina 将这些化合物对接至该口袋。这些数据用于微调 CHEM-BERT 模型,以从化合物的 SMILES 字符串预测对接分数。之后,使用微调后的模型扫描整个 Enamine HLL-460 化合物库(46 万个化合物),然后对预测分数最佳的 10,000 个化合物再次使用 AutoDock Vina 进行对接,以获得实际对接分数。为评估模型预测准确性,展示了所选前 10,000 个分子中 Vina 分数与预测分数的相关性。
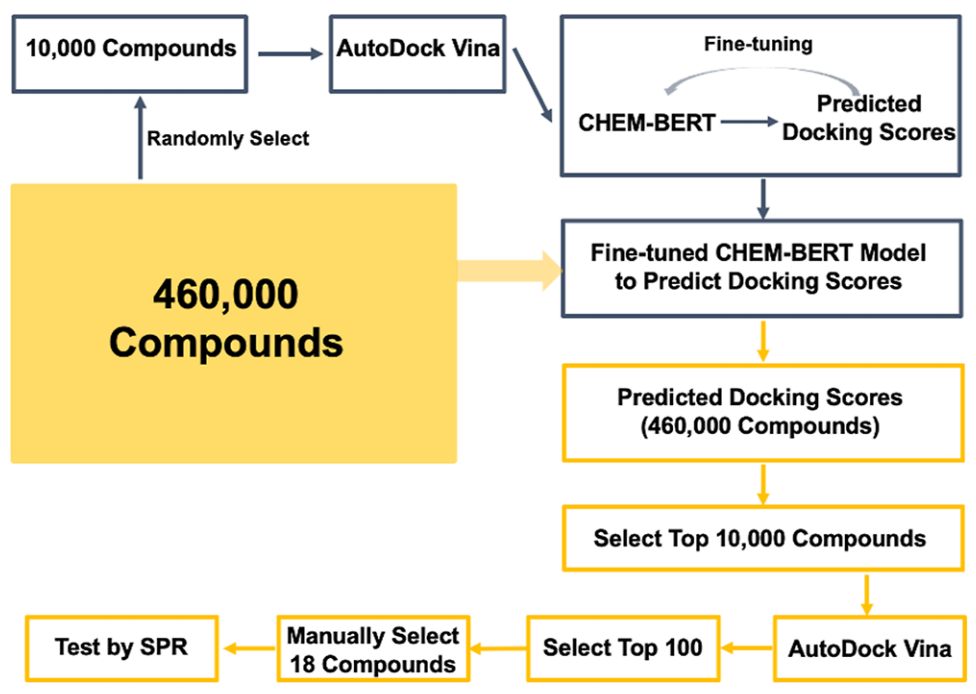
从对接分数前 100 的化合物列表中,通过目视检查对接模式筛选出 18 个化合物并从
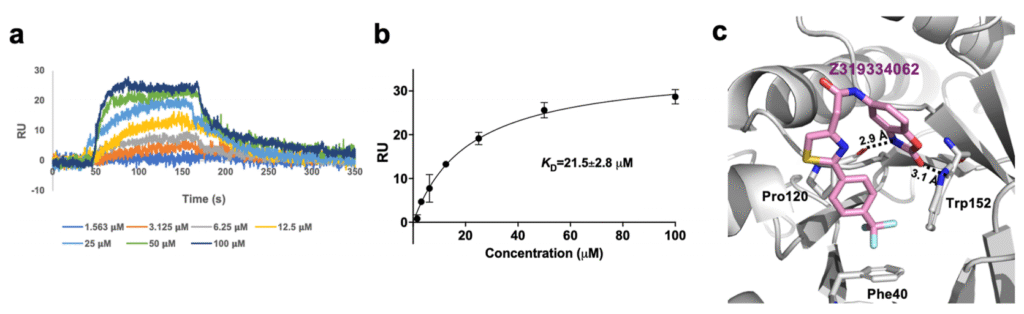
Enamine进行采购。其中,7 个化合物未显示任何表面等离子体共振(SPR)结合信号,10 个化合物显示非特异性相互作用,1 个化合物 Z319334062 对 PRMT5 N 端 TIM Barrel结构域显示出特异性结合亲和力(KD=21.5μM)(图 5a、b)。Z319334062 的 Vina 分数为-8.1 千卡/摩尔。同时,通过 200ns 的分子动力学模拟观察相互作用细节。苯并[d]噁唑-2 (3H)-酮在分子一侧与 Pro120 主链和 Trp152 侧链形成两个氢键以稳定结合,而另一侧的(三氟甲基)苯则嵌入由 Phe40、Val83、Pro120、Ala121 和 Trp152 侧链组成的疏水口袋中。中间的酰胺键和噻唑作为连接体连接两部分(图5c)。通过 MM-PBSA 估算的不含熵的结合能为-8.7±3 千卡 / 摩尔。
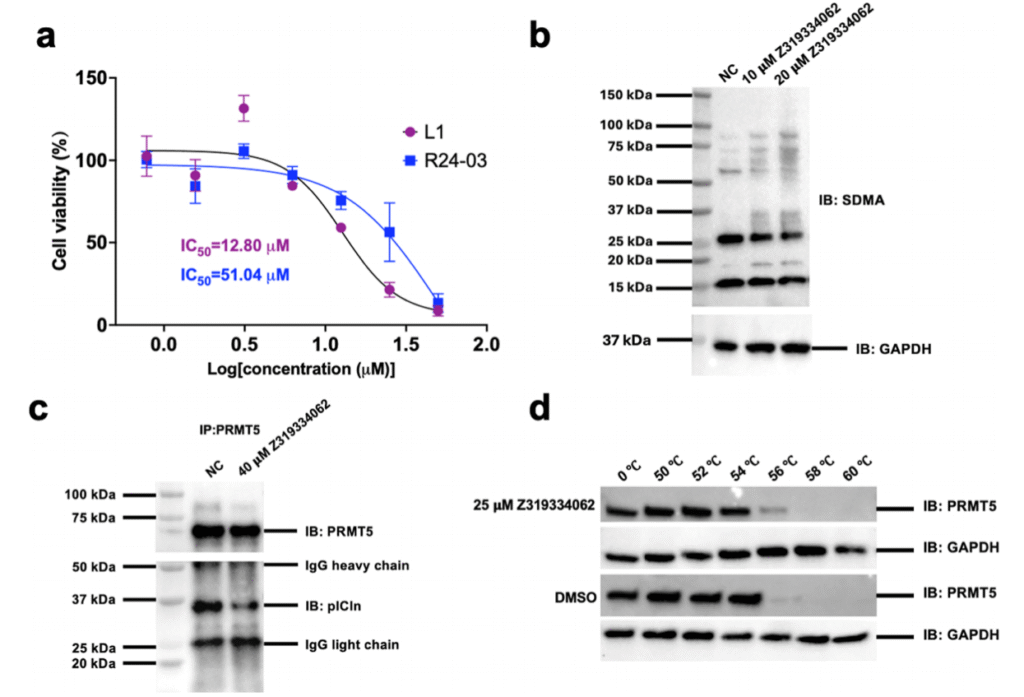
为确定测试化合物细胞活性的细胞系,分析了六种肿瘤类型的 RNA 测序数据,发现只有胶质母细胞瘤(GBM)的肿瘤组织中 PRMT5 和 pICLn 表达水平较高。因此,为评估 Z319334062 在细胞环境中是否能破坏 PRMT5/pICln 相互作用,选择了两种 PRMT5 和 CLNS1A 表达水平不同的患者来源胶质母细胞瘤(GBM)细胞系 L1 和 R24-03。在细胞活力测定中,处理 4 天后,L1 细胞的半数抑制浓度(IC50=12.8μM)显著低于 R24-03 细胞(IC50=51.0μM),表明 PRMT5/CLNS1A 过表达的细胞系对药物更敏感(图 6a)。此外,蛋白质印迹法显示,用 Z319334062 和 GSK3326595(阳性对照)处理细胞后,对称性二甲基化精氨酸(SDMA)水平降低,表明 PRMT5 甲基转移酶功能受到抑制(图 6b)。研究发现,Z319334062 处理仅抑制部分底物,这与McKinney等人发表的数据一致。最后,为测试 Z319334062 是否能破坏 L1 细胞中 PRMT5 与 pICLn 的相互作用,进行了共免疫沉淀实验。结果显示,与阴性对照相比,40μM Z319334062 处理后,PRMT5/pICLn 复合物被破坏(图 6c)。同时,25μM Z319334062 处理 18 小时后,PRMT5 的熔解温度显著变化(图 6d)。
结论
本研究应用基于机器学习的计算机虚拟筛选方法识别潜在的 PRMT5 N 端 TIM Barrel结合配体,随后对 18 种潜在抑制剂进行生化筛选,其中一种被认为是有前景的非共价 PRMT5/pICLn 抑制剂。在本次筛选中,效率提高了约 96%。具体而言,使用基于机器学习的流程,仅需对接 20,000 个化合物(共 39,090 个变体 = 22,191 个训练变体 + 16,889 个所选化合物变体)。相比之下,对接整个库需要处理 460,000 个化合物(共 867,859 个变体)。此外,5.6% 的命中率虽适中,但相较于高通量筛选中通常低于 1% 的命中率有显著提升。由于 PRMT5 的 N 端结构域是新发现的药物结合位点,目前仅报道了两种抑制剂与其结合:一种是共价结合剂 BRD0639(KD=13.8μM),另一种是环肽肽 50(KD=89±11nM),两者均经过多轮优化发现。相比之下,本研究的先导化合物仅通过基于机器学习的虚拟筛选,未经过任何优化,就显示出约 20μM 的结合亲和力,凸显了其巨大潜力。
为观察结合情况,对活性化合物 Z319334062 与 TIM Barrel结构域结合的状态进行了 200ns 的分子动力学模拟,以分析结合模式,为进一步的构效关系优化提供依据。此外,通过癌症基因组图谱(TCGA)数据库筛选,选择了两种代表性的 GBM 细胞系进行细胞研究。结果表明,在剂量依赖性处理下,Z319334062 在 PRMT5/CLNS1A 过表达的细胞系中效果更显著。此外,Z319334062 在细胞水平上也能破坏 PRMT5/pICLn 相互作用。这些证据表明,Z319334062 可能是设计更有效的新型癌症治疗类似物的先导化合物。
分子对接
使用 Schrodinger 蛋白质制备协议在 pH7.4±2.0 下制备靶标(PDB:7SER),删除与配体原子形成少于两个氢键的水分子。使用 Schrodinger Ligprep 处理配体,从 SMILES 字符串生成三维结构,以及所有可能的质子化状态和立体异构体,产生 22,191 个 “变体”。 Grid Box以 pICln(残基 205-234)为中心,各方向为 14.0Å。所有对接均使用 AutoDock Vina 1.2 进行,exhaustiveness程度为 32。在分析和机器学习模型训练中,仅考虑每个分子的最佳对接分数。
机器学习模型
使用样本库的 SMILES 字符串和对接分数微调 CHEM-BERT 模型。训练集按 80:10:10 的比例分为训练集、验证集和测试集。仅使用训练集和验证集对模型进行 14 个 epoch 的训练,选择第 4 个 epoch 获得的模型(验证损失最低)。在预留的测试集上预测的分数与实际 Vina 分数的相关性为 r²=0.77,使用该最终模型预测整个库的 Vina 分数。
分子动力模拟
Z319334062 与 PRMT5 N 端(1-292)结合的初始结构来自虚拟筛选步骤。将系统放置在截断的八面体盒子中,盒子边距距任何溶质原子至少 20Å。用 OPC 水溶剂化系统,用 Na + 和 Cl - 离子中和电荷,并添加额外离子使盐浓度达到 0.15M。蛋白质原子使用 Amber ff19SB 力场,Z319334062 配体使用 GAFF 力场表示。然后对系统进行 100 步 minimization(仅允许水移动),随后进行 10,000 步 minimization(允许所有原子移动)。系统在恒定体积下加热 0.6ns 至 310K,然后在最终温度下松弛 0.4ns。随后在最终温度和恒定 1atm 压力下松弛 2ns,最后在相同条件下进行 200ns 模拟。

